
肿瘤微环境(TME)调节着肿瘤的生存、维持、生长和免疫监测,在疾病进展和治疗反应中起着重要作用,如局部耐药、免疫逃逸和癌症转移等。阐明不同细胞群中TME细胞的组成及其功能有助于探索更有效的肿瘤治疗方法。

为了解卷积稀有的细胞类型,研究团队对代表41个亚群的386个样本进行了FACS分类和测序。最终收集的数据集和样本被注释为18个TME细胞类型和41个存在于血液中的细胞群,共有51个独特的细胞群。(图1H)
通过收集全面纯化细胞群的RNA,研究团队重建了类似组织和血液的批量RNA图谱,并通过人为地混合来自不同纯化细胞亚群的RNA来模仿组织的异质性,以创建数百万个人工肿瘤转录组,用于训练Kassandra。(图1J)
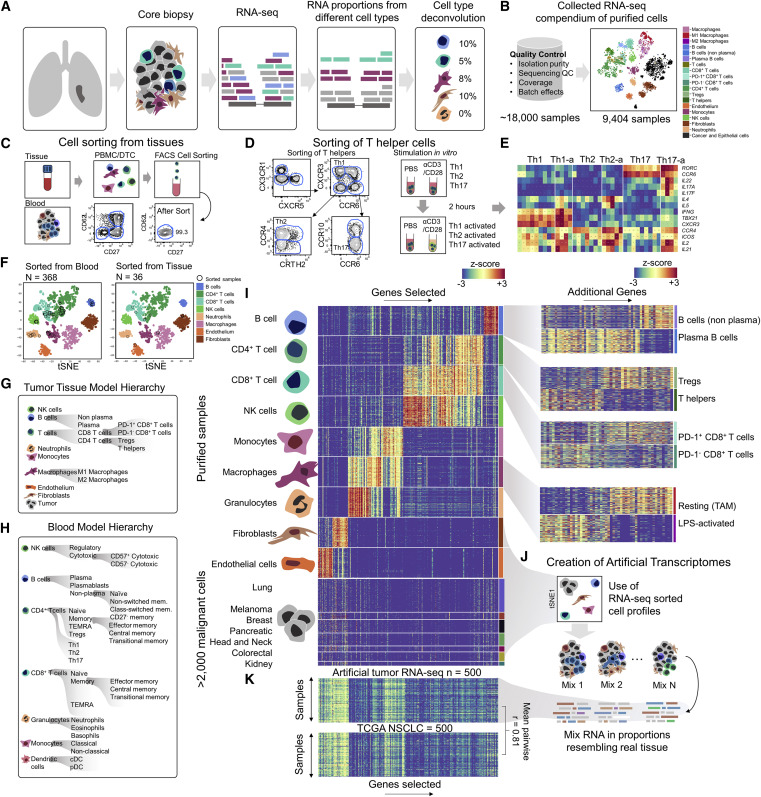
图1. 定义不同分类细胞群的RNA图谱,人工重建组织。
Kassandra使用每百万份reads的转录本(TPM)作为表达单位。通常,属于短RNA转录本总表达的可变性会强烈扭曲相关基因的TPM值分布(图2)。非编码RNA以及转录组中标注为V、D或J区域对应的T细胞受体(TCR)和B细胞受体(BCR)编码基因的短转录本被TPM归一化排除在外;由于基因表达计算中的噪声增加,低转录本支持水平且未验证的转录本和部分未知编码序列的转录本也被排除在归一化之外。
为此,研究团队重新定义了先进的TPM归一化,降低了管家基因的变化影响。转录本过滤和TPM再规范化使基因表达在不同数据集之间更具可比性。主成分分析(PCA)显示,TPM归一化减少了分类细胞RNA图谱汇编中不同数据集的表达批次效应。
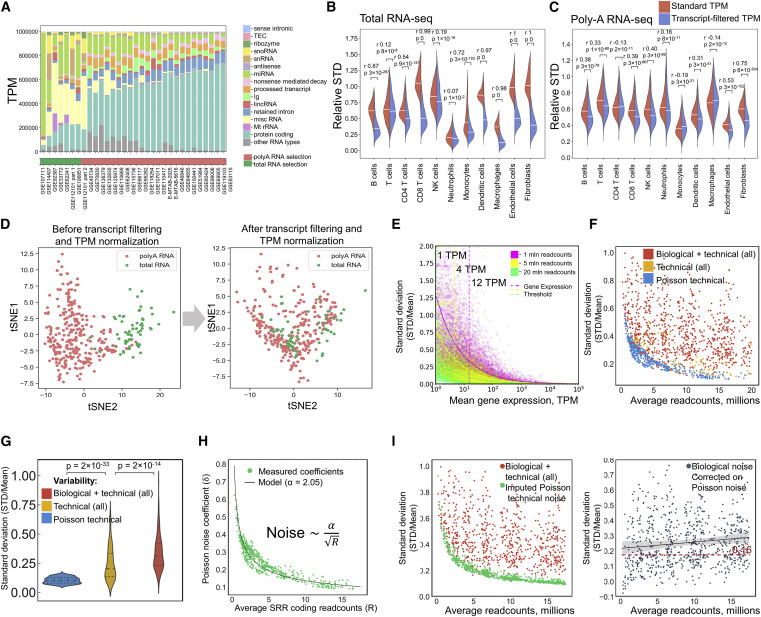
图2. 表达式归一化的建立和技术噪声分析。
由于可用的具有已知细胞组成的TME RNA-seq数据集十分有限,研究团队开发了组织和血液的人工RNA-seq,用于训练Kassandra算法,以稳健地识别不同的细胞群。所有可用的分类细胞群和癌症细胞系的组合被生成,以构建癌症特异性的人工转录组,模仿生物变异性。最终,共产生1800万和800万转录组,分别用于训练Kassandra-Tumor和Kassandra-Blood模型。(图3)
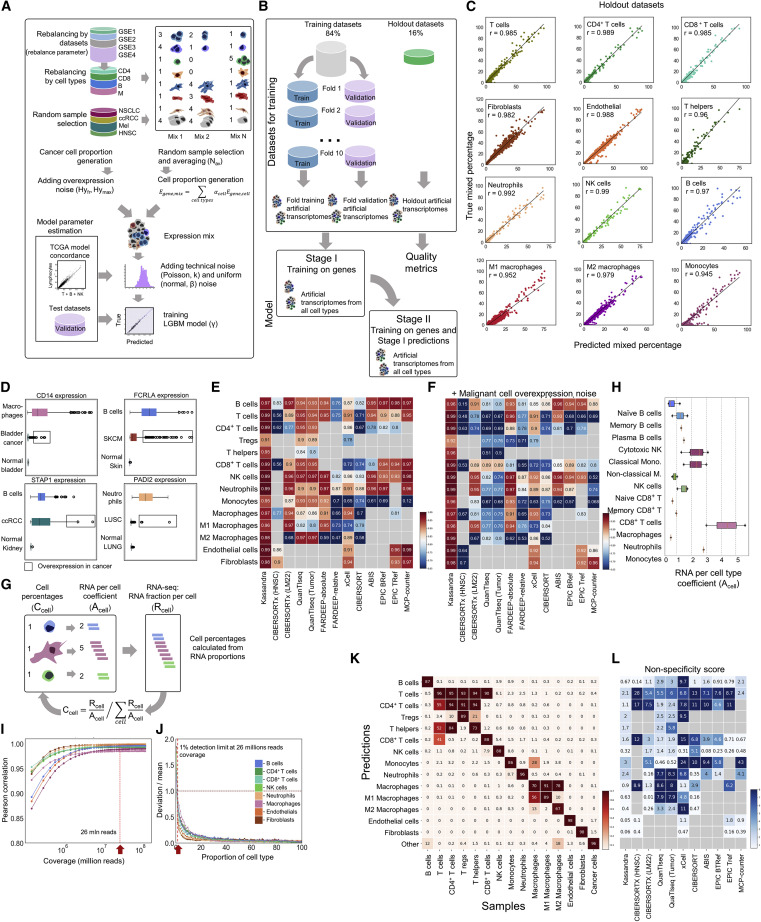
图3. 人工转录组上Kassandra反卷积的训练和表现。
Kassandra经开发和训练后,研究团队使用Kassandra-Tumor分析了不同TCGA肿瘤和健康组织的细胞组成(图4)。结果显示,胰腺腺癌中成纤维细胞比例最高,含有大量癌症相关成纤维细胞(CAFs);巨噬细胞是影响预后和治疗的关键因素,据预测,在多形性胶质母细胞瘤和肺腺癌的TME中,巨噬细胞占10%或更多。当将Kassandra应用于基因型-组织表达(GTEx)数据集时,单个核细胞主要在血液样本中发现。上述结果表明,在预期范围内,Kassandra反卷积准确识别、推算了不同组织中的细胞类型。
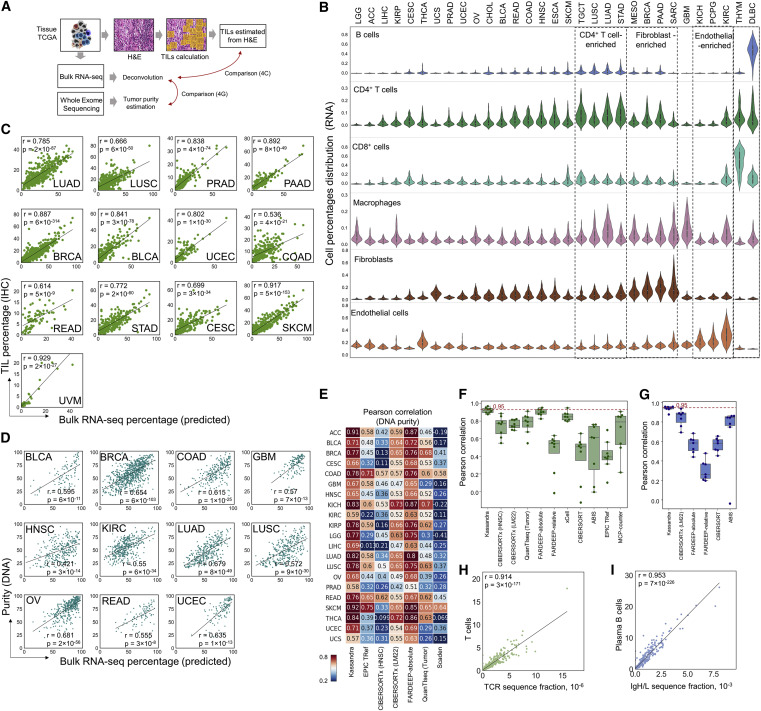
图4. 健康组织和肿瘤组织RNA-seq的大规模TME反卷积。
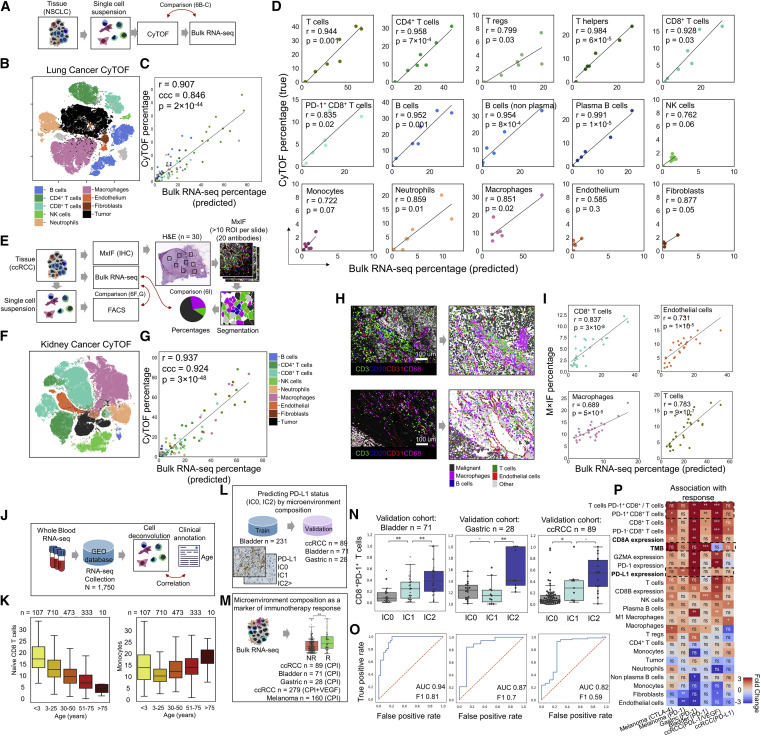
进一步,研究团队在临床环境中收集了不同级别的原发性早期非小细胞肺癌(NSCLC)和透明细胞肾细胞癌(ccRCC)肿瘤,测试了Kassandra准确重建TME的能力。研究团队对NSCLC、ccRCC肿瘤标本进行批量RNA-seq和CyTOF分析,并使用40多种细胞标记对为CyTOF制备的相同细胞悬液进行RNA-seq。(图5)
结果显示,与其他方法相比,Kassandra与T和B细胞、中性粒细胞、巨噬细胞、Treg、NK细胞、内皮细胞和成纤维细胞群的CyTOF检测具有较强的相关性。t-SNE分析显示,只有免疫细胞(ICs)能从肿瘤样本中得到有效回收,Kassandra准确预测了ccRCC样本中的免疫群体,且相关性最强。
综上所述,研究团队开发了基于决策树ML的Kassandra算法,使用批量RNA-seq重建肿瘤活检和血液中的细胞组成,并收集了一个包含不同细胞群9,400多个分类样本的RNA-seq数据库,用于创建人工转录组以训练、开发Kassandra。基于其独特的RNA特征,Kassandra算法可应用于不同细胞类型,可使人们更好、更全面地了解仅含RNA-seq数据的档案样本中的生物学,未来有望支持多种疾病的临床应用。
参考文献:
Aleksandr Zaitsev, Maksim Chelushkin, Daniiar Dyikanov. et al. Precise reconstruction of the TME using bulk RNA-seq and a machine learning algorithm trained on artificial transcriptomes. Cancer cell (2022). DOI:https://doi.org/10.1016/j.ccell.2022.07.006
声明:本文来源测序中国,仅为交流学习。内容仅代表作者个人观点,望大家理性判断及应用。













