肝癌的发病率和死亡率在癌症中居前列,全球每年新诊断病例超过90万,死亡人数超过80万。90%的肝癌病例是肝细胞癌 (HCC),病人的存活率在很大程度上取决于诊断时的疾病分期。当肿瘤处于定植期(localized),五年生存率为34%,占患者总数的44%;侵袭期(regional)五年生存率为12%,占患者总数的27%;发现远处的病灶时即转移期(distant),患者的五年生存率为3%,占患者总数18%。全世界有3.5亿人患有慢性病毒性肝炎感染,5000万人患有肝硬化,这些人群的肝癌风险增加。高风险人群的筛查目前采用带或不带甲胎蛋白 (AFP) 的腹部超声成像,筛查灵敏度从47%-84%不等,特异性从67%-90%以上不等。因此,非常需要开发可用且灵敏的非侵入性HCC筛查方法。
近日,美国约翰霍普金斯大学研究团队开发出一种基于血液的全基因组cfDNA片段化特征检测方法,为HCC检测提供了一种高性能且具有成本效益的可行选择。该研究结果发表在Cancer Discovery上,题为“Detecting liver cancer using cell-free DNA fragmentomes”。研究人员分析了HCC患者中cfDNA的分子来源,并确定了与片段化变化相关的基因组和染色质特征。
研究人员检测了501名个体的血浆样本,来源于75名HCC患者和426名非癌症患者。在非癌症的个体中,133人患有会增加HCC风险的疾病,包括各种原因引起的肝硬化或无肝硬化的病毒性肝炎。研究团队利用0.5-5ml血浆生成基因组文库,并对cfDNA片段进行低覆盖率全基因组测序(~2.6倍覆盖率),每个样本平均有4900万个高质量配对reads,包含9Gb的序列数据。
同时,研究团队还检测了223名来自香港患者的全基因组序列数据作为验证队列,包括可切除的早期HCC((n=90, a期85,B期5)、HBV (n=66)和乙肝相关肝硬化(n=35),以及无肝病的健康个体(n=32)。
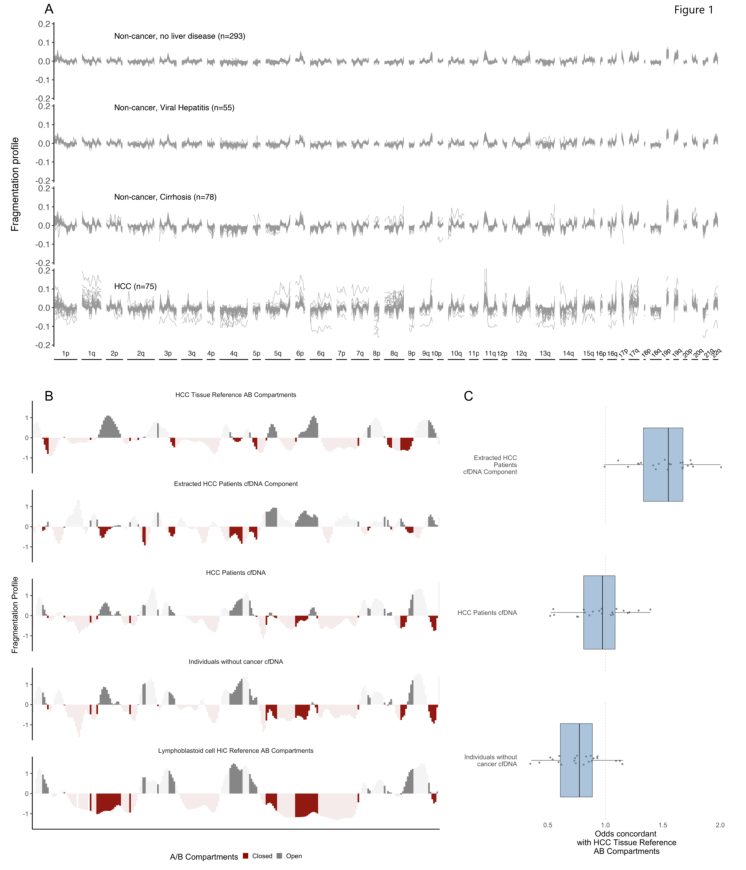
研究人员评估了cfDNA片段谱并在473个非重叠的5MB区域中生成了整个基因组的片段谱,每个区域包含约80,000个片段,并使用DELFI方法跨越大约2.4GB的基因组。结果显示,cfDNA片段谱在无癌个体中是一致的,HCC患者中存在较大的差异(图1A)。 与HCC患者相比,肝硬化患者的特征更接近于未患肝硬化的非癌症个体(图1A)。病毒性肝炎患者的片段谱与无肝病的非癌症个体几乎相同(图1A)。
为了研究cfDNA片段化模式的起源,研究人员将全基因组片段组图谱与高通量测序染色体构象捕获(Hi-C)开放(A)和封闭(B)区室进行了比较,发现健康个体的cfDNA片段化模式与淋巴母细胞高度相关(图 1B)。相比之下,无癌个体的概况更接近淋巴母细胞的A/B区室(图1B、C)。以上分析表明,来自HCC患者的cfDNA片段组代表了外周血细胞和肝癌细胞染色质区室的cfDNA图谱的混合。
图1. 全基因组片段谱反映了潜在的染色质结构。来源:Cancer Discov
3.从全基因组cfDNA片段推断疾病特异性转录因子
研究团队分析了cfDNA片段化特征是否可能反映了肝癌中转录因子(TF)的DNA结合改变引起的变化。研究团队比较了HCC患者与无癌个体的TF,以确定在cfDNA中全基因组结合位点覆盖率差异最大和最小的TF(图2A、B)。使用基因疾病关联的DisGeNET数据库进行的基因集富集分析显示,预计HCC和无癌个体之间的cfDNA TF结合覆盖率差异与HCC和其他癌症相关(图2C、D)。对HCC患者的cfDNA片段化数据进行类似分析,显示与HCC相关的转录因子结合位点的覆盖范围差异有所增加(图2C、E)。以上结果表明,HCC和其他癌症患者的cfDNA片段化变化是由癌细胞中存在的大量转录谱改变引起的。
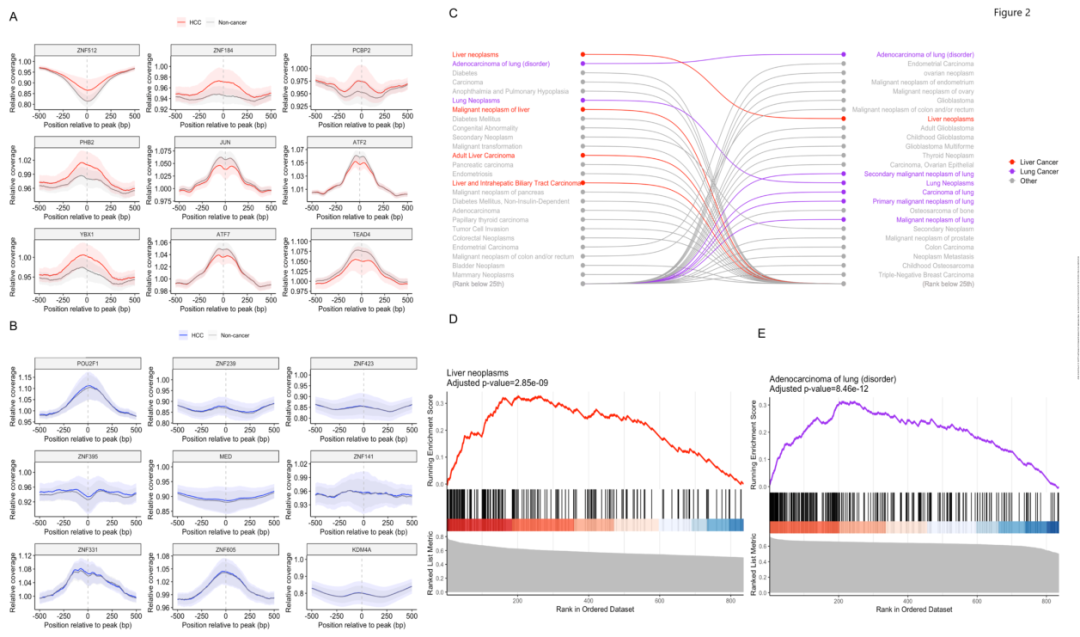
图2. HCC患者的片段化谱突出了肝脏特异性转录因子。来源:Cancer Discov
3.DELFI利用cfDNA片段组揭示HCC的基因组变化
鉴于HCC的基因组和染色质变化与cfDNA片段化之间的直接联系,研究人员使用机器学习方法确定了cfDNA片段组的变化是否可以区分HCC患者和非癌症患者,构建了DELFI模型。结果显示该机器学习模型,检测癌症的敏感性在平均风险人群中为88%,特异性为98%,在高危人群中为85%,特异性为80%。除了利用在HCC患者中观察到的染色质和转录因子变化导致的全基因组片段化谱外(图3A),该模型分析还揭示了与HCC中常见的获得或丢失的匹配染色体臂的表征改变,例如之前报道的HCC的TCGA大规模基因组研究(n = 372)(图3B)。
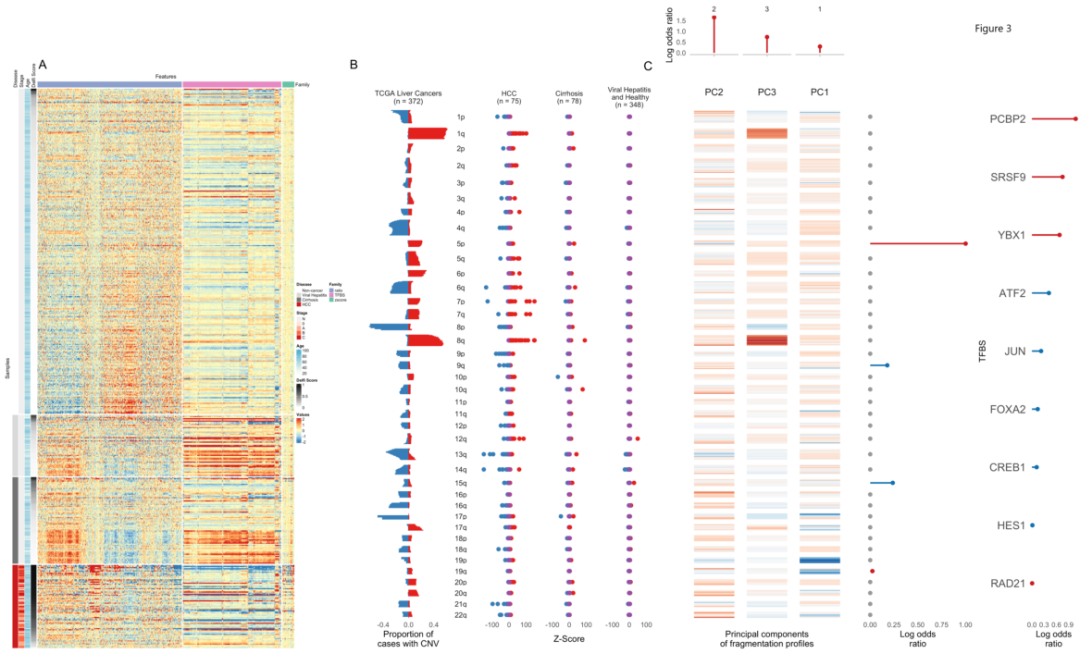
图3. 片段化特征反映了HCC生物学,并被纳入DELFI机器学习方法中。来源:Cancer Discov
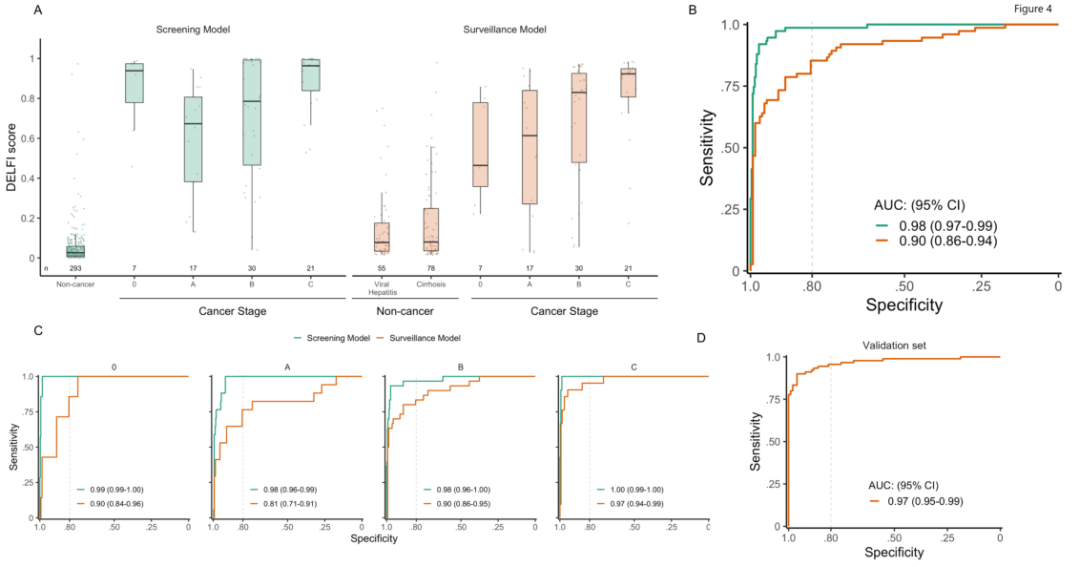
接下来,研究人员检查了DELFI评分与高危人群中HCC发生和分期之间的关系。133名非癌症个体的DELFI分数较低,病毒性肝炎或肝硬化患者的DELFI分数中位数分别为0.078或0.080。相比之下,75名HCC患者在所有阶段的DELFI中位数得分显著更高(图4A)。用于识别HCC患者的DELFI方法的特征(ROC)曲线显示,在高风险个体中,曲线下面积(AUC)为0.90(图4B)。早期HCC的表现仍然稳健,晚期HCC个体几乎完全被检测到(AUC>0.97)(图4C)。在亚洲人验证队列中,DELFI模型可将AUC为0.97的HCC患者与高危个体区分开来(图 4D),表明cfDNA片段化的基本特征在该队列中相似,并且DELFI是检测HCC的可靠方法,或可在不同高危人群中推广。
图4.DELFI机器学习模型以高灵敏度和特异性检测HCC。来源:Cancer Discov
研究团队开发的全基因组cfDNA片段化特征检测方法对HCC具有高灵敏度和特异性。研究表明片段化概况捕获了基因组和染色质特征,包括已知在HCC中很重要的改变。cfDNA片段化组分析方法“DELFI”是第一个在单独的高风险人群中独立验证的全基因组片段化分析,在检测HCC方面具有稳定和强大的性能,包括非常早期的疾病,且与疾病病因无关。
该研究结果还表明,疾病特异性转录因子特征可以通过全基因组cfDNA片段谱进行分析。利用全基因组cfDNA片段分析疾病特异性转录调控,可能改善癌症患者起源组织的检测和识别。随着患者数量的增加,cfDNA转录谱可以进一步改进机器学习算法来检测HCC和其他癌症。
Foda, Z. H., Annapragada, A. V., Boyapati, K., Bruhm, D. C., Vulpescu, N. A., Medina, J. E., ... & Velculescu, V. E. Detecting liver cancer using cell-free DNA fragmentomes. Cancer Discovery,(2022). https://doi.org/10.1158/2159-8290.CD-22-0659
https://aacrjournals.org/cancerdiscovery/article/doi/10.1158/2159-8290.CD-22-0659/711014/Detecting-liver-cancer-using-cell-free-DNA
声明:本文来源测序中国,仅为交流学习。内容仅代表作者个人观点,望大家理性判断及应用。